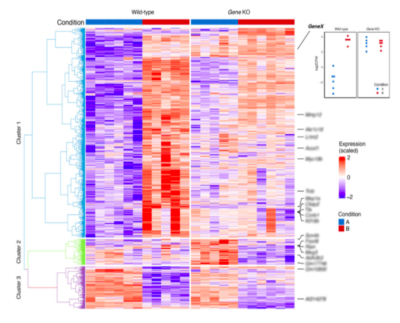
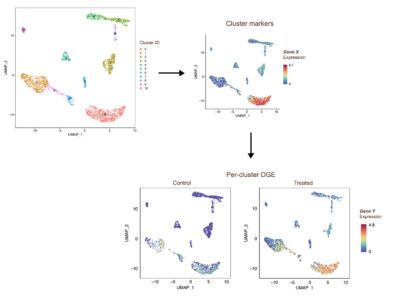
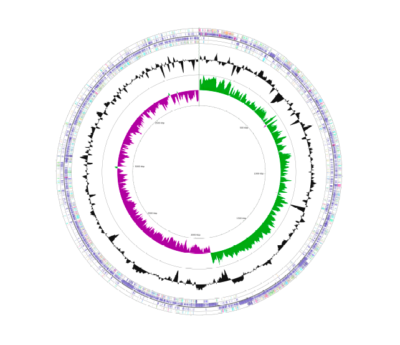
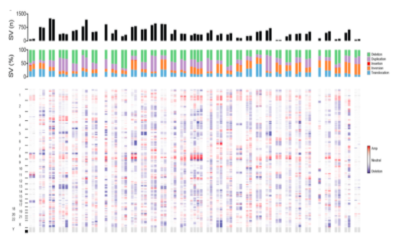
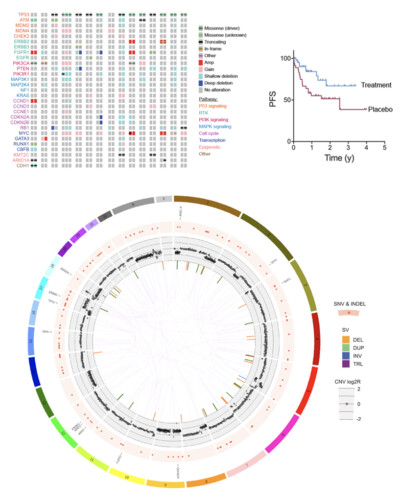
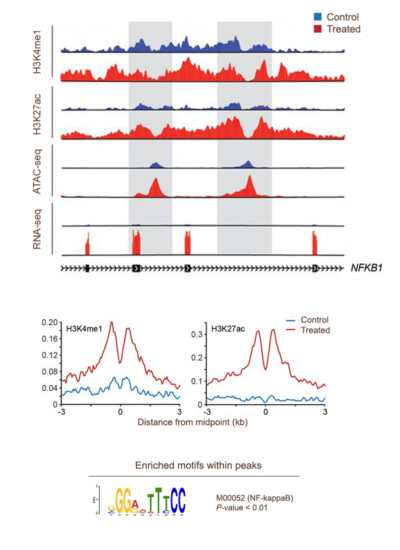
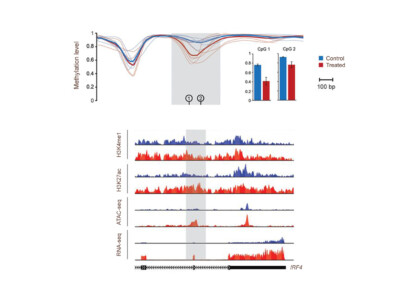
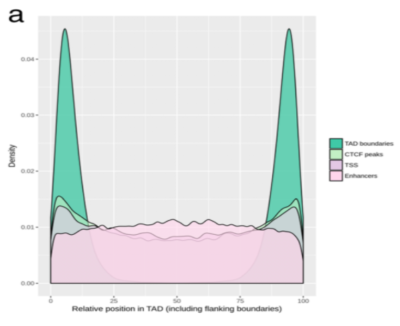
Le C3G possède une expérience significative dans l’analyse et l’organisation de données provenant de différents essais biologiques, en exploitant les méthodes de calcul de pointe. Nous offrons également une large gamme de services d’analyse standardisée et personnalisée aux chercheurs, qui bénéficient à leur tour de notre expérience dans l’analyse de données provenant de diverses applications de séquençage. Nous pouvons vous offrir notre expertise dans les domaines énumérés ci-dessous:
-
- Analyse de vos données ou données publiques par nos bio-informaticiens experts
- Planifier une expérience avec un fournisseur de séquençage
- Soutien pour les demandes de subvention de collaboration.
- Service de formation
- Développer un pipeline d’analyse de données personnalisé
- Organiser et distribuer les données
Depuis 2015, nous avons offert nos services dans différents domaines de la bio-informatique à de nombreux chercheurs, institutions et entreprises.
Demandes Communes
Partenaires de la Plateforme (Services des Wet-Lab)
Partenaires de la Plateforme (Services des Wet-Lab)

Le Centre de génomique McGill offre aux chercheurs canadiens et internationaux des technologies à haut débit et des approches de pointe pour soutenir les études génomiques de nouvelle génération. Les initiatives menées par nos scientifiques nous ont permis d’acquérir des technologies de pointe et d’intégrer des méthodologies novatrices pour façonner l’avenir de la génomique. En Savoir plus

Le Centre d’expertise et de services (CES) offre à la communauté scientifique des services de première qualité dans le domaine de séquençage (y compris le séquençage parallèle), de génotypage, de génomique fonctionnelle et d’extraction d’acides nucléiques. Ils fournissent des services complets d’analyse de l’ADN et de l’ARN, à partir de quelques échantillons jusqu’à plusieurs dizaines de milliers par semaine et travaillent en étroite collaboration avec Le Centre canadien de génomique computationnelle(C3G) pour offrir des services de bio-informatique. En Savoir plus

Le GCRC a développé une série de plateformes d’innovation dotées d’une technologie de pointe, chacune est gérée et composée d’un personnel dévoué et hautement qualifié. Ces plateformes d’innovation sont essentielles à la recherche menée par nos membres et sont également accessibles à d’autres chercheurs de McGill et à des collaborateurs externes. Veuillez noter qu’en cliquant sur le lien, vous serez redirigé vers une page d’information sur le GCRC qui est uniquement disponible en anglais. Nous nous excusons pour tout inconvénient.